Theory8. K-Means Clustering and Gaussian Mixture Model(2)
8. K-Means Clustering and Gaussian Mixture Model(2)
\(\newcommand{\argmin}{\mathop{\mathrm{argmin}}\limits}\) \(\newcommand{\argmax}{\mathop{\mathrm{argmax}}\limits}\) Machine Learning의 기초적인 이론부분을 다시 제대로 잡고 싶어서 문일철 교수님의 인공지능 및 기계학습 개론을 정리한 Post입니다.
- 8.3 Multinomial Distribution
- 8.4 Mutivariate Gaussian Distribution
- 8.5 Gaussian Mixture Model
8.3 Multinomial Distribution
- Selecting 0 or 1 => Binomial Distribution
- Selecting 0 or 1 or …. or M => Multinomial Distribution
주사위 던지기 예제에 적용하여 생각해 보자.
- x: 선택지 ex) 주사위한번 던졌을 경우 숫자가 나온 개수(0,0,1,0,0,0)
- \(\mu\): 확률 ex) 주사위의 각각의 숫자가 나올 확률
$$\sum_{k}x_k = 1, P(X|\mu) = \prod_{k=1}^{K} \mu_k^{x_k}, \mu_k \ge 0, \sum_{k}\mu_{k}=1$$
위의 식을 살펴보게 되면 Binormial Distribution의 General한 Version인 것을 확인할 수 있다.
주사위의 6가지 경우의 수가 아닌 실제 Dataset에 적용시키면 다음과 같이 식을 변형할 수 있다.
- D: Dataset with N selections => \(x_1, ..., x_n\)
$$P(X|\mu) = \prod_{n=1}^{N} \prod_{k=1}^{K} \mu_k^{x_{nk}} = \prod_{k=1}^{K} \mu_k^{\sum_{n=1}^{N} x_{nk}} = \prod_{k=1}^{K} \mu_k^{m_k}$$
ex) 주사위를 25번 던졌다.
- x: x1, x2, …, x25
- k: 1,2,3,4,5,6
- \(\mu\): 1/6, 1/6, 1/6, 1/6, 1/6, 1/6
MLE of Multinormial Distribution
Multinormial Distribution의 확률을 구할 수 있으므로, 이 확률의 MLE는 다음과 같이 나타낼 수 있다.
$$\text{Maximize} P(X|\mu) = \prod_{k=1}^{K} \mu_{k}^{m_k}$$
$$\text{Subject to} \mu_k \ge 0, \sum_{k} \mu_k = 1$$
어떠한 Function을 Maximize하는데 제약조건이 있으므로 5. SVM과 같이 Largrange Method로서 해결한다.
$$L(\mu, m, \lambda) = \sum_{k}m_k ln(\mu_k) + \lambda(\sum_{k}\mu_k -1)$$
$$\frac{d}{d \mu_k}L(\mu, m, \lambda) = \frac{m_k}{\mu_k} + \lambda =0 \rightarrow \mu_k = -\frac{m_k}{\lambda}$$
$$\sum_{k} \mu_k =1 \rightarrow -\sum_{k}\frac{m_k}{\lambda} = 1 \rightarrow \sum_{k} m_k = -\lambda \rightarrow \sum_{k}\sum_{n}x_{nk} = N$$
$$\therefore -\lambda = N \rightarrow \mu_{k} = \frac{m_k}{N}$$
MLD of Multinormial Distribution은 MLE of Bionomial Distribution(\(\hat{\theta} = \frac{\alpha_H}{\alpha_H+\alpha_T}\))와 형태가 같은 \(\frac{\text{해당 횟수}}{\text{전체 횟수}}\)라는 것을 확인할 수 있다.
8.4 Multivariate Gaussian Distribution
먼저 Gaussian Distribution의 식을 다시한번 생각해보면 다음과 같이 나타낼 수 있다.
$$N(x|\mu,\sigma^2) = \frac{1}{\sqrt{2\pi \sigma^2}}exp(-\frac{1}{2\sigma^2}(x-\mu)^2)$$
위와 같은 식은 \(\mu, \sigma\)가 각각의 1개의 값을 가지는 단순한 Gaussian Distribution이다.
위의 식을 활용하여 Multivariabe Gaussian Distribution을 나타내면 다음과 같이 나타낼 수 있다.
$$N(x|\mu,\boldsymbol{\Sigma}) = \frac{1}{2\pi^{D/2}}\frac{1}{|\boldsymbol{\Sigma}|^{1/2}}exp(-\frac{1}{2}(x-\mu)^T \boldsymbol{\Sigma}^{-1}(x-\mu))$$
\(\mu, \boldsymbol{\Sigma}\)이 더이상 단일 값이 아니라 Dimension의 개수만큼 가져야 하므로, Matrix로서 표현하게 되어서 식이 변하게 되었다.
\(\boldsymbol{\Sigma}\)은 Covariance Matrix로서 표현된다.
실제 잘 감이 안잡히므로 Multivariate Gaussian Distribution을 Visualization하면 다음과 같다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import cm
from mpl_toolkits.mplot3d import Axes3D
# Our 2-dimensional distribution will be over variables X and Y
N = 60
X = np.linspace(-3, 3, N)
Y = np.linspace(-3, 4, N)
X, Y = np.meshgrid(X, Y)
# Mean vector and covariance matrix
mu = np.array([0., 1.])
Sigma = np.array([[ 1. , -0.5], [-0.5, 1.5]])
# Pack X and Y into a single 3-dimensional array
pos = np.empty(X.shape + (2,))
pos[:, :, 0] = X
pos[:, :, 1] = Y
def multivariate_gaussian(pos, mu, Sigma):
"""Return the multivariate Gaussian distribution on array pos.
pos is an array constructed by packing the meshed arrays of variables
x_1, x_2, x_3, ..., x_k into its _last_ dimension.
"""
n = mu.shape[0]
Sigma_det = np.linalg.det(Sigma)
Sigma_inv = np.linalg.inv(Sigma)
N = np.sqrt((2*np.pi)**n * Sigma_det)
# This einsum call calculates (x-mu)T.Sigma-1.(x-mu) in a vectorized
# way across all the input variables.
fac = np.einsum('...k,kl,...l->...', pos-mu, Sigma_inv, pos-mu)
return np.exp(-fac / 2) / N
# The distribution on the variables X, Y packed into pos.
Z = multivariate_gaussian(pos, mu, Sigma)
# Create a surface plot and projected filled contour plot under it.
fig = plt.figure()
ax = fig.gca(projection='3d')
ax.plot_surface(X, Y, Z, rstride=3, cstride=3, linewidth=1, antialiased=True,
cmap=cm.viridis)
cset = ax.contourf(X, Y, Z, zdir='z', offset=-0.15, cmap=cm.viridis)
# Adjust the limits, ticks and view angle
ax.set_zlim(-0.15,0.2)
ax.set_zticks(np.linspace(0,0.2,5))
ax.view_init(27, -21)
plt.show()
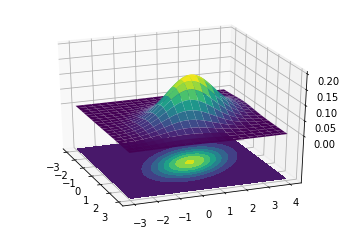
Mixture Model
이러한 Gaussian Mixture Model을 사용해야 하는 이유를 생각하면 다음과 같다.
현재 다음과 같은 Data Distribution이 있다고 하자.

위와 같은 상황인 경우 Gaussian Distribution으로서 Data Distribution을 예측하면 다음과 같다.

잘 Fitting이 되지 않는다고 할 수 있다.
즉, Multivariabe Gaussian Distribution로서 여러개의 평균과 분산으로서 Gaussian Distribution여러개를 사용하여 Data Distribution을 Prediction하면 다음과 같은 형태가 된다.

위와 같은 이유로서 GMM(Gaussian Mixture Model)을 사용하는 것 이다.
이러한 GMM을 식으로서 표현하면 다음과 같다.
$$P(x) = \sum_{k=1}^{K} \pi_{k} N(x|\mu_k,\sigma_k)$$
$$\sum_{k=1}^{K}\pi_k =1, 0 \le \pi_k \le 1$$
중요하게 봐야하는 점은 \(\pi_k\)은 K개의 Gaussian Distribution에서 각각의 Gaussian Distribution의 속할 확률로서 0~1 사이의 값을 가지게 된다. 이는 K-Means의 Hard Clustering(Cluster에 속하냐 안하냐로서만 표현- 0 or 1)이 아닌, Soft Clustering으로서 표현된다는 것 이다.
참조(Covariance Matrix)
Covariance Matrix는 정말 많이 사용되는 Matrix의 종류 중 하나이므로 꼭 이해하고 넘어가야 한다.
각각의 Element에 관하여 Covariance 관계를 나타낸 Matrix이고 수학적인 수식으로 나타내면 다음과 같다.
위의 Covariance Matrix에서 주요하게 봐야하는 점은 4가지 이다.
- Covariance Matrix는 Symmentric Matrix 이다.
- Covariane Matrix의 Diagonal은 각각의 Element의 Variance을 의미한다.
- Covariance Matrix의 Diagonal을 제외한 각각의 Element는 두개의 Element끼리의 Covariance를 의미한다.
Covariance Matrix를 좀 직관적으로 살피기 위하여 2차원이라고 한정하고 몇개의 예시를 살펴보면 다음과 같다.
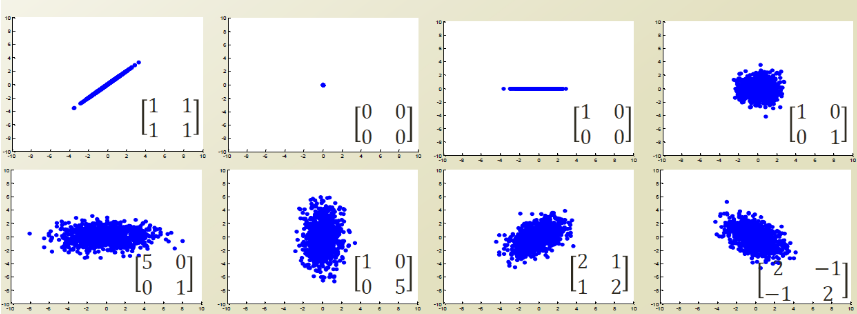
위의 그림을 왼쪽 위부터 살펴보면 다음과 같다.
- Varaince와 Covariance가 각각 1이다. 즉, x축으로의 분산, y축으로의 분산이 각각 1이고, Correlation값을 계산항여도 1이니, y=x와 같은 형태로서 나타내게 된다.
- Variance와 Covariance가 각각 0 이다. 즉, x축,y축으로의 분산이 없는 상태이므로 하나의 Point로서 나오게 된다.
- x축으로만 분산이 있는 상황이다. 즉, 2차원에서 x축으로만 값이 존재하는 형태로서 표현된다.
- x,y축으로의 분산만 존재하는 상황이다. Covariance가 0이므로 원의 형태로서 나오게 된다.
- x축의 분산이 y축의 분산보다 큰 상황이다. 즉 타원형태이지만 x축의 길이가 더 길다.
- 5와 형태가 같다.
- 1의 형태에서 분산만 두배로 커진다.
- 7의 형태에서 Covariance의 부호만 바뀌게 된다. => Correlation의 방향이 반대이다.
위와 같은 Covariance의 특징을 통하여 SVD, PCA에서도 사용하게 된다.
이전 Post SVD의 식을 살펴보면 다음과 같다.
$$A = U \sum V^{T}$$
위의 식을 다시한번 생각해보게 되면, Orthogonal Matrix(U,V)로서 회전변환 하여 Covariance Matrix에서 Diagonal형태가 되도록 EigenVector를 설정한다. Eigen Vector에서 값이 적은 축을 제거 한다. => 위의 예시에서 3번인 경우 y축 제거
위와 같은 PCA를 그림으로 살펴보면 다음과 같다.
사진 참조: https://medium.com/mighty-data-science-bootcamp/unsupervised-learning-pca-k-means-a95aa72bf27f
8.5 Gaussian Mixture Model
$$P(x) = \sum_{k=1}^{K} \pi_{k} N(x|\mu_k,\sigma_k) = \sum_{k=1}^{K}P(z_k)P(x|z)$$
$$\sum_{k=1}^{K}\pi_k =1, 0 \le \pi_k \le 1$$
- \(\pi_{k} = P(z_k)\): 어떤 Cluster를 선택할 것 인가. Mixing Coefficient
- \(N(x|\mu_k,\sigma_k)=P(x|z)\): Cluster의 Gaussian Distribution Mixture Component
Example) 실제 Data x가 들어왔을 때 어떤 Cluster에 Assign될 것인가?
$$\gamma(z_{nk}) = P(z_k=1|x_n) = \frac{P(z_k=1)P(x|z_k=1)}{\sum_{j=1}^{K}P(z_j=1)P(x|z_j = 1)}= \frac{\pi_k N(x|\mu_k,\boldsymbol{\Sigma}_k)}{\sum_{j=1}^{K}\pi_j N(x|\mu_j \boldsymbol{\Sigma}_j)}$$
위의 식은 매우 간단하다.
MLE of Multiormial Distribution와 같이 \(\frac{해당 Cluser에 속할 확률}{전체 Cluster에 속할 확률}\)로서 표현하게 된다.
위의 식(Gaussian Mixture Model)을 Log Likelihood로서 표현하면 다음과 같이 나타낼 수 있다.
$$ln(P(X|\pi,\mu,\boldsymbol{\Sigma})) = \sum_{n=1}^{N}ln[\sum_{k=1}^{K} \pi_k N(x|\mu_k,\boldsymbol{\Sigma}_k)]$$

![{\displaystyle \operatorname {K} _{\mathbf {X} \mathbf {X} }={\begin{bmatrix}\mathrm {E} [(X_{1}-\operatorname {E} [X_{1}])(X_{1}-\operatorname {E} [X_{1}])]&\mathrm {E} [(X_{1}-\operatorname {E} [X_{1}])(X_{2}-\operatorname {E} [X_{2}])]&\cdots &\mathrm {E} [(X_{1}-\operatorname {E} [X_{1}])(X_{n}-\operatorname {E} [X_{n}])]\\\\\mathrm {E} [(X_{2}-\operatorname {E} [X_{2}])(X_{1}-\operatorname {E} [X_{1}])]&\mathrm {E} [(X_{2}-\operatorname {E} [X_{2}])(X_{2}-\operatorname {E} [X_{2}])]&\cdots &\mathrm {E} [(X_{2}-\operatorname {E} [X_{2}])(X_{n}-\operatorname {E} [X_{n}])]\\\\\vdots &\vdots &\ddots &\vdots \\\\\mathrm {E} [(X_{n}-\operatorname {E} [X_{n}])(X_{1}-\operatorname {E} [X_{1}])]&\mathrm {E} [(X_{n}-\operatorname {E} [X_{n}])(X_{2}-\operatorname {E} [X_{2}])]&\cdots &\mathrm {E} [(X_{n}-\operatorname {E} [X_{n}])(X_{n}-\operatorname {E} [X_{n}])]\end{bmatrix}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/595ae6dc8ee7f0708dbf854a48a8c6bfad7ff8ce)
Leave a comment