R-분류분석
분류(Classfication)
소속 집단을 알고 있는 데이터를 이용하여 모형을 만들어서 소속집단을 모르는 데이터들의 집단을 결정하는 기법
Supervised Learning.
- 로지스틱 회귀
자세한내용 - 의사결정 나무(Decision Tree)
- 랜덤 포레스트(Random Forest)
- 나이브베이즈 분류(Naive Bayes Classification)
- SVM(Support Vector Machine)
- K-NN Classfication
의사결정 나무(Dicision Tree)
의사결정나무분석은 탐색과 모형화라는 두 가지 특징을 가지고 있다. 의사결정나무분석은 판별분석, 회귀분석 등과 같은 모수적(parameter) 모형을 분석하기 위해 사전에 이상치(outlier)를 검색하거나 분석에 필요한 변수 또는 모형에 포함되어야할 상호작용의 효과를 찾아내기 위해서 사용될 수도 있고, 의사결정나무 자체가 분류 또는 예측모형으로 사용될 수도 있다.
참조: dreamlog 블로그
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
#DecisionTree
#데이터 분류 - Train, Test
idx<-sample(1:nrow(iris),nrow(iris)*0.7)
train<-iris[idx,]
test<-iris[-idx,]
#Model 생성
formula<-Species~Sepal.Length+Sepal.Width+Petal.Length+Petal.Width
iris_ctree<-ctree(formula = formula,data=train)
iris_ctree
#Model 확인
plot(iris_ctree)
#Test Data 예측
pred<-predict(iris_ctree,test)
#정확도 예측 - 1
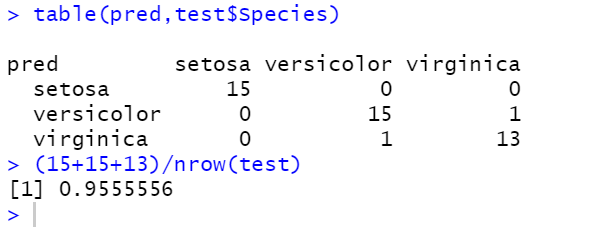
table(pred,test$Species)
(15+15+13)/nrow(test) #정확도: 0.955...
#정확도 예측 - 2
install.packages("caret")
install.packages("e1071")
library(caret)
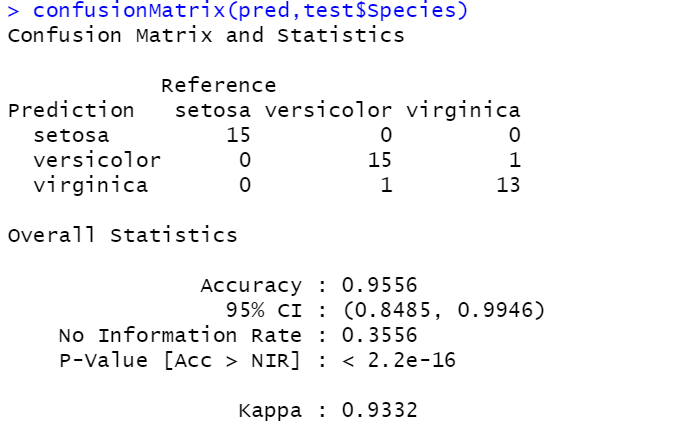
confusionMatrix(pred,test$Species)
의사결정나무 그림:
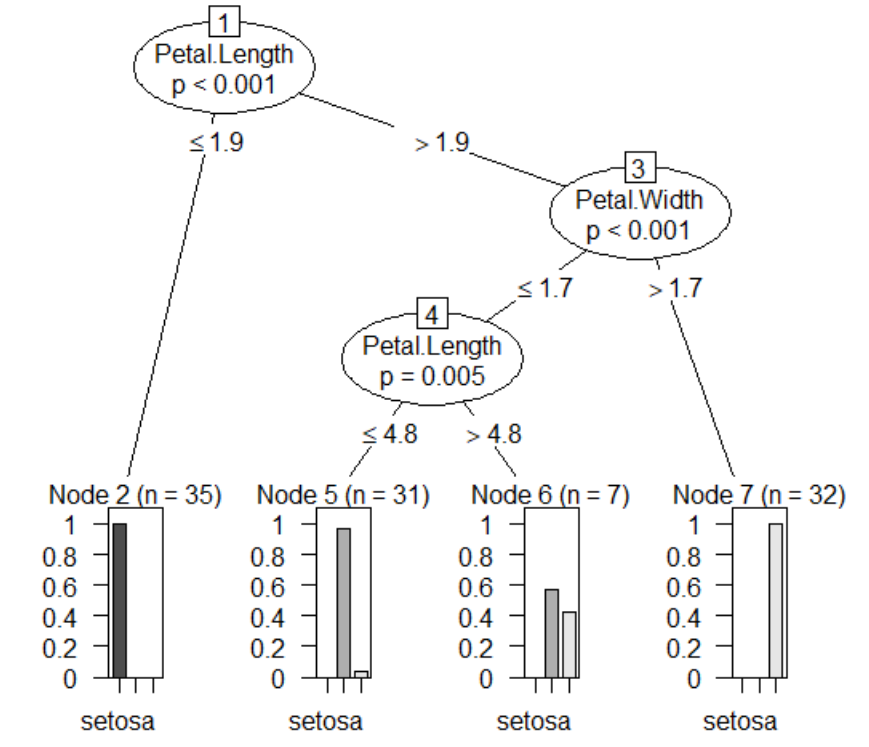
Petal.Length, Petal.Width 순으로 Species에 영향을 미친다는 것을 알 수 있다.
정확도 확인1:
정확도 확인2:
랜덤 포레스트(Random Forest)
랜덤포레스트는 분류, 회귀 분석 등에서 사용되는 앙상블 학습 기법의 일종으로, 훈련 과정에서 구성한 다수의 결정 트리로부터 분류 또는 평균예측치를 출력함으로써 동작한다.
의사결정 나무를 여러개 합쳐놓은 형태 이다.
의사결정 나무에 비해 분류 정확도는 높으나 속도는 다소 떨어진다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
#Random Forest
install.packages("randomForest")
library(randomForest)
#Data 나누기 - Train,Test
set.seed(123)
n<-sample(2,nrow(iris),replace = T,prob=c(0.7,0.3))
n
train<-iris[n==1,]
test<-iris[n==2,]
dim(train)
dim(test)
#Model 만들기
model<-randomForest(Species~.,data=train)
model
#Model 정확도 측정
(32+28+33)/nrow(train) # Accuracy: 0.93
#Model2 만들기 - 결측치 제거
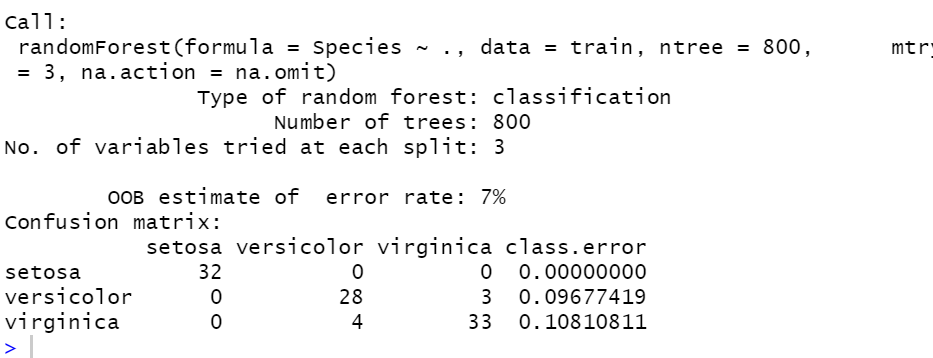
model2<-randomForest(Species~.,data=train,ntree=800,mtry=3,na.action = na.omit)
model2
#Model2 정확도 측정
(32+28+33)/nrow(train) #Accuracy: 0.93
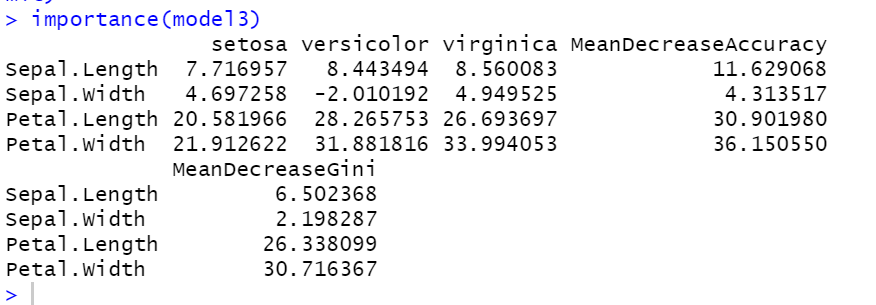
#중요 독립변수 확인
model3<-randomForest(Species~.,data=train,importance=T,na.action = na.omit)
importance(model3)
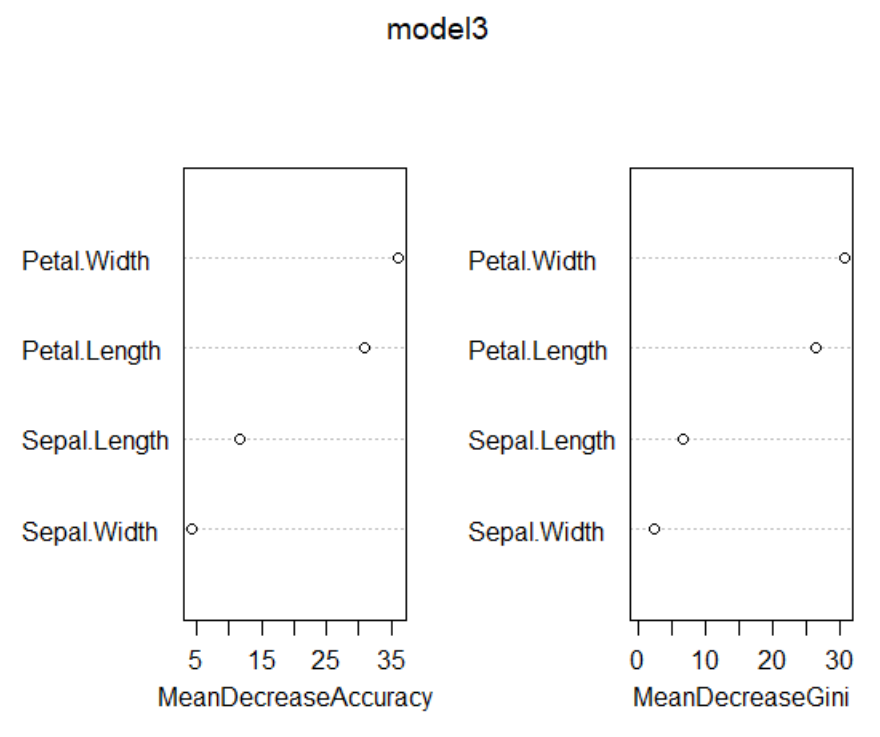
varImpPlot(model3)
#Peta.Width -> Petal.Length 수준으로 중요도가 있는것을 확인 할 수 있다.
pred<-predict(model3,test)
t<-table(pred,test$Species)
t
#Accuracy: 0.98
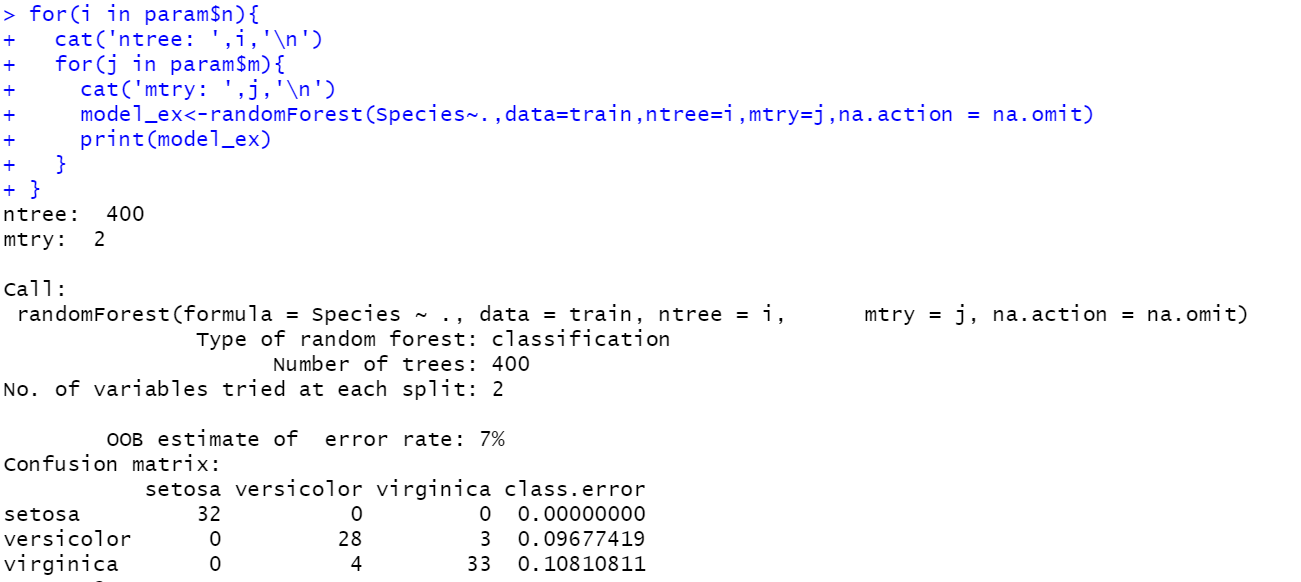
#가장좋은 Tree의 갯수값 구하기
ntree<-c(400,500,600)
mtry<-c(2:4)
param<-data.frame(n=ntree,m=mtry)
param
for(i in param$n){
cat('ntree: ',i,'\n')
for(j in param$m){
cat('mtry: ',j,'\n')
model_ex<-randomForest(Species~.,data=train,ntree=i,mtry=j,na.action = na.omit)
print(model_ex)
}
}
#ntree: 400, mtry:2 가 가장 좋은 결과를 얻었다.
#ntree와 mtry의 값은 해보기 전에 알 수 없어 for 구문을 돌려 알아봐야 한다.
랜덤포레스트 Model1:

랜덤포레스트 Model2:
랜덤포레스트 Model3 - 독립변수 중요도:
랜덤포레스트 Model3-그림:
랜덤포레스트 Model3-정확도:

랜덤포레스트 ntree, mtry 구하기:
나이브베이즈 분류(Naive Bayes Classification)
나이브 베이즈 정리
조건부 확률 P(A|B)는 사건 B가 발생할 경우 A의 확률을 나타낸다.
베이즈 정리는 P(A|B)의 추정이 P(AnB)와 P(B)에 기반을 두어야 한다는 정리이다.

예제

전체 사건 중 비가 온 확률은 P(비) = 7/20 입니다. 그렇다면 비가 안온 확률은 얼마인가?
P(~비) = 13/20 이겠죠. 비가 오는지 안오는지 같이 둘 중 하나의 상태만 가능한 사건들은 모든 경우의 수를 더했을 때 1이 된다.
그렇다면 이제 P(비|맑은날) 의 값은 얼마인가?

위 식을 통해 P(비|맑은날) 을 구하기 위해선 P(맑은날|비), P(비), P(맑은날) 이 세개의 값만 알아내면 된다.

P(비|맑은날) = P(맑은날|비) * P(비) / P(맑은날)
= (2/7) * 0.35 / 0.5 = 0.2
전체중에서 맑은날 이면서 비가올 확률은 20% 정도 된다고 볼 수 있다.
참조:Gom Guard 블로그
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
#나이브베이지안(NaiveBayes Classfication)
library(e1071)
set.seed(123)
#ham,spam을 분류
#Data 가져오기
sms_data<-read.csv("https://raw.githubusercontent.com/pykwon/Test-datas-for-R/master/sms_spam_tm.csv")
sms_data
names(sms_data)
dim(sms_data)
sms_data[1,c(1:10)]
head(sms_data$sms_type)
sms_data_df<-sms_data[-1]
sms_data_df
str(sms_data_df)
#Data 분류하기
set.seed(123)
idx<-sample(nrow(sms_data_df),nrow(sms_data_df)*0.7)
train<-sms_data_df[idx,]
test<-sms_data_df[-idx,]
nrow(train)
nrow(test)
#Model 만들기
model_sms<-naiveBayes(sms_type~.,data=train)
#Model로 예측하기
pred_sms<-predict(model_sms,test,type="class")
pred_sms
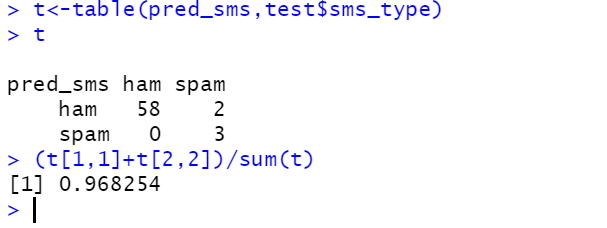
#정확도 측정정
t<-table(pred_sms,test$sms_type)
t
(t[1,1]+t[2,2])/sum(t)#정확도: 0.968...
결과:
SVM(Support Vector Machine)
SVM이란 원 훈련(또는 학습)데이터를 비선형 매핑을 통해 고차원으로 변환한다. 이 새로운 차원에서 초평면(Hyperplane)을 최적으로 분리하는 찾는다. 즉, 최적의 Decision Boundary(의사결정 영역)을 찾는다
SVM의 목적은 Margin을 최대화 하는 Decision을 찾는 것. Margin: Hyperplne을 기준으로 plus-plane, minus-plane와의 거리.
참조:Excelsior-JH 블로그
참조:Ratsgo 블로그
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
#SVM
#데이터 불러오기
attach(iris)
#독립변수 종속변수 선언
x<-subset(iris,select = Species)
y<-Species
#SVM Model 만들기
svm_model<-svm(Species~.,data = iris)
summary(svm_model)
#SVM 시각화 하기
par(mar = rep(2, 4))
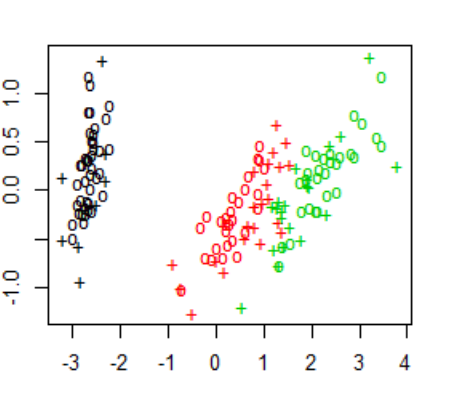
plot(cmdscale(dist(iris[,-5])),col=as.integer(iris[,5])
,pch=c("o","+")[1:150%in%svm_model$index+1])
SVM Model 결과:

SVM Model 시각화:
K-NN Classfication
K-NN 알고리즘은 지도학습(Supervised Learning)의 한 종류로 레이블이 있는 데이터를 사용하여 분류 작업을 하는 알고리즘이다. 알고리즘의 이름에서 볼 수 있듯이 데이터로부터 거리가 가까운 K개의 다른 데이터이 레이블을 참조하며 분류하는 알고리즘이다. 주로 거리를 측정할 때 유클리디안 거리 계산법을 사용하여 거리를 측정하는데, 벡터의 크기가 커지면 계산이 복잡해진다.
장점
- 알고리즘이 간단하여 구현하기 쉽다.
- 수치 기반 데이터 분류 작업에서 성능이 좋다.
단점
- 학습 데이터의 양이 많으면 분류 속도가 느려진다.
- 차원의 크기가 크면 계산량이 많아진다.
참조:PROINLAB 블로그
유클리디안 거리

참조:Nick Lib
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
#K-NN
#필요한 패키지 설치
install.packages("ggvis")
library(ggvis)
#데이터 분포 시각화
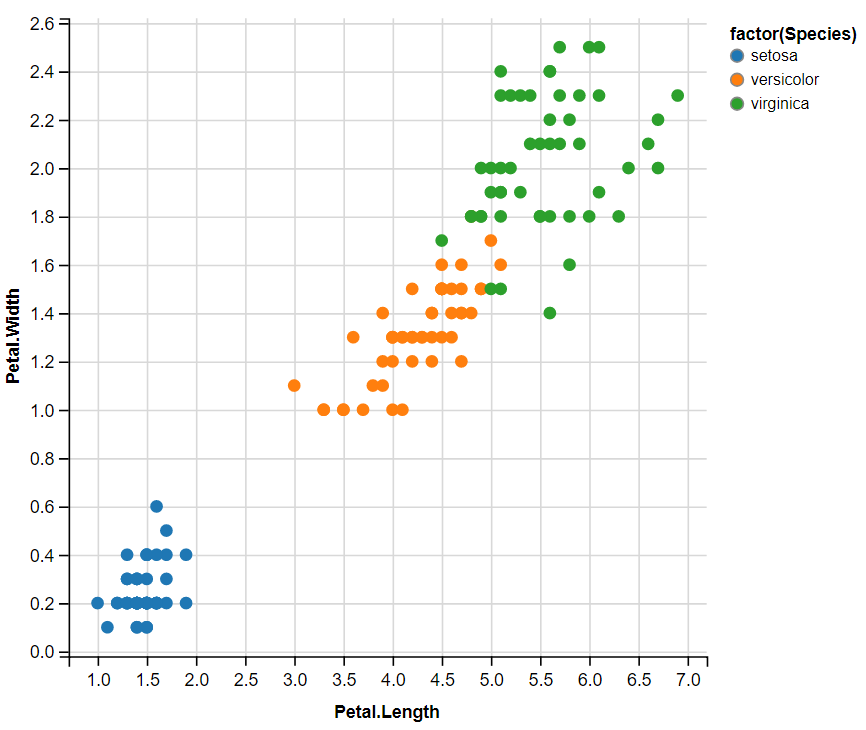
iris%>%ggvis(~Petal.Length,~Petal.Width,fill=~factor(Species))
#정규화 과정: 정규화란 전체 구간을 0~100으로 설정하여 데이터를 관찰하는 방법
#(요소값-최소값)/(최대값-최소값)
func_normal<-function(x){
num<-x-min(x)
m_n<-max(x)-min(x)
return (num/m_n)
}
test_df<-data.frame(x=c(1:5))
test_df
func_normal(test_df)
#데이터 전처리 과정(정규화및 DataFrame으로 만들기)
lapply(iris[1:4], func_normal)
normal_d<-as.data.frame(lapply(iris[1:4], func_normal))
head(normal_d)
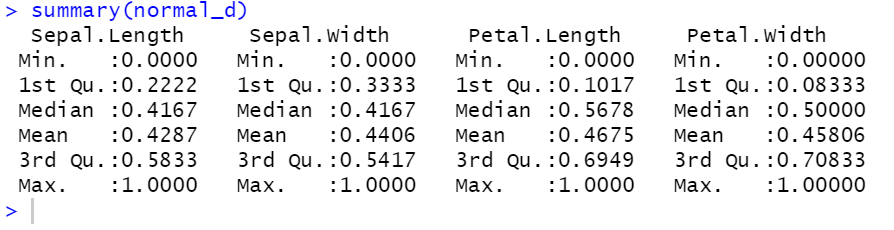
summary(normal_d)
df<-data.frame(normal_d,Species=iris$Species)
head(df)
#Train, Test Data로 나누기
set.seed(123)
idx<-sample(1:nrow(df),0.7*nrow(df))
ir_train<-df[idx,]
ir_test<-df[-idx,]
nrow(ir_train):nrow(ir_test)
#Model 만들기
library(class)
model<-knn(train=ir_train[,-5],test=ir_test[,-5]
,cl=ir_train$Species,k=3)
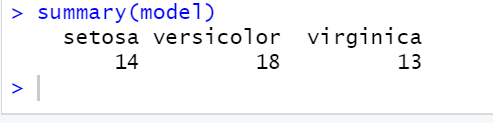
summary(model)
#Model 정확도 측정
t<-table(model,ir_test$Species)
t
(t[1,1]+t[2,2]+t[3,3])/nrow(ir_test)
변수 분포 시각화::
변수 정규화 결과:
Model 결과:
Model 정확도:

잠조: 원본코드
코드에 문제가 있거나 궁금한 점이 있으면 wjddyd66@naver.com으로 Mail을 남겨주세요.

Leave a comment