Paper28. Multimodal Dynamics: Dynamical Fusion for Trustworthy Multimodal Classification
Multimodal Dynamics: Dynamical Fusion for Trustworthy Multimodal Classification
- Paper: Multimodal Dynamics: Dynamical Fusion for Trustworthy Multimodal Classification
- Code: mmdynamics Github
Abstract
Integration of heterogeneous and high-dimensional data (e.g., multiomics) is becoming increasingly important. Existing multimodal classification algorithms mainly focus on improving performance by exploiting the complementarity from different modalities. However, conventional approaches are basically weak in providing trustworthy multimodal fusion, especially for safety-critical applications (e.g., medical diagnosis). For this issue, we propose a novel trustworthy multimodal classification algorithm termed Multimodal Dynamics, which dynamically evaluates both the feature-level and modality-level informativeness for different samples and thus trustworthily integrates multiple modalities. Specifically, a sparse gating is introduced to capture the information variation of each within-modality feature and the true class probability is employed to assess the classification confidence of each modality. Then a transparent fusion algorithm based on the dynamical informativeness estimation strategy is induced. To the best of our knowledge, this is the first work to jointly model both feature and modality variation for different samples to provide trustworthy fusion in multi-modal classification. Extensive experiments are conducted on multimodal medical classification datasets. In these experiments, superior performance and trustworthiness of our algorithm are clearly validated compared to the state-of-the-art methods.
해당 논문에서는 dynamically evaluates both the feature-level and modality-level informativeness 할 수 있는 “Multimodal Dynamics”라는 Model을 제시한다. 이러한 Modeld의 장점으로는 True class probability를 사용하여 Feature와 Modality의 Confidence을 측정할 수 있고 이로 인하여 Multimodality간의 trustworhily integrate를 수행할 수 있다는 것 이다.
Introduction
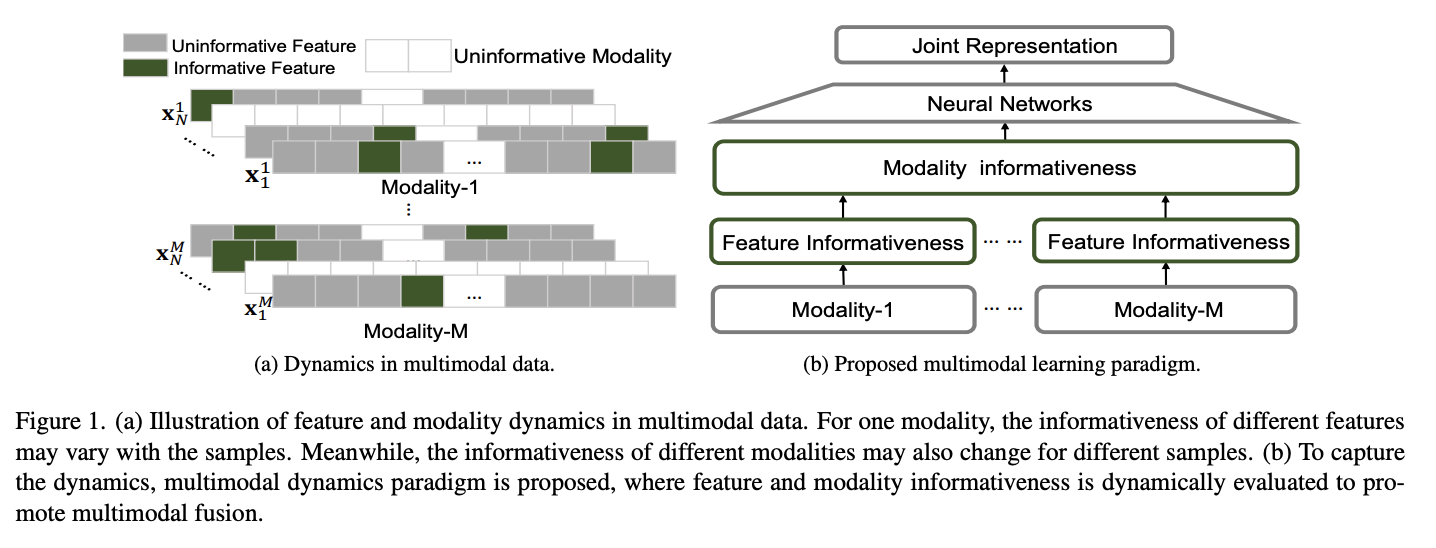
For multimodal learning, traditional methods mainly focus on obtaining a common or joint representation by exploring the correlated and complementary information between different modalities with powerful neural networks. Although effective, these methods are weak in dynamically perceiving the informativeness of each feature and modality for different samples, which could enhance the trustworthiness (including stability and explainablity) in multimodal classification. In this work, we propose a novel algorithm termed Multimodal Dynamics for trustworthy multimodal classification, which models the feature and modality informativeness to promote the fusion stability and explainablity. Specifically, we introduce a sparse gating strategy to dynamically obtain the informative features for different samples.
Bio Domain에서 Multi-modality를 사용하기 위한 문제점은 개인적으로 생각할 때, 크게 3가지 이다. (1) Low sample, (2) Data Imbalance, (3) High Dimensional Dataset, (4) Data heterogeneous, (5) Unmatches sample dataset (equal to 1).
해당 논문은 이러한 문제점중에서 2, 5을 제외한 모든 문제를 해결하기 위한 Modeld인 “Multimodal Dynamics”을제안한다.
먼저 위의 Figure 1(a)를 보게 되면, Bio Domain에서는 각각의 Modality가 High Dimension이므로 중요한 feature (informative feature)를 뽑는것이 중요하다.
이러한 과정을 제시하는 model에서는 “Feature Informativeness”를 통하여 해결 하였다.
또한, 많은 BioData에서 각각의 Modality의 중요도는 다르다. 예를 들어 Alzheimer’s Disease를 예측하는데 있어서 PET, sMRI는 효과적이나 Gene expressiond은 performance가 낮은 것을 알 수 있다. 이러한 문제점을 해결하기 위하여 해당 논문에서는 “Modality Informativeness”를 추가하였다 (Figure 1(b)).
Proposed Method
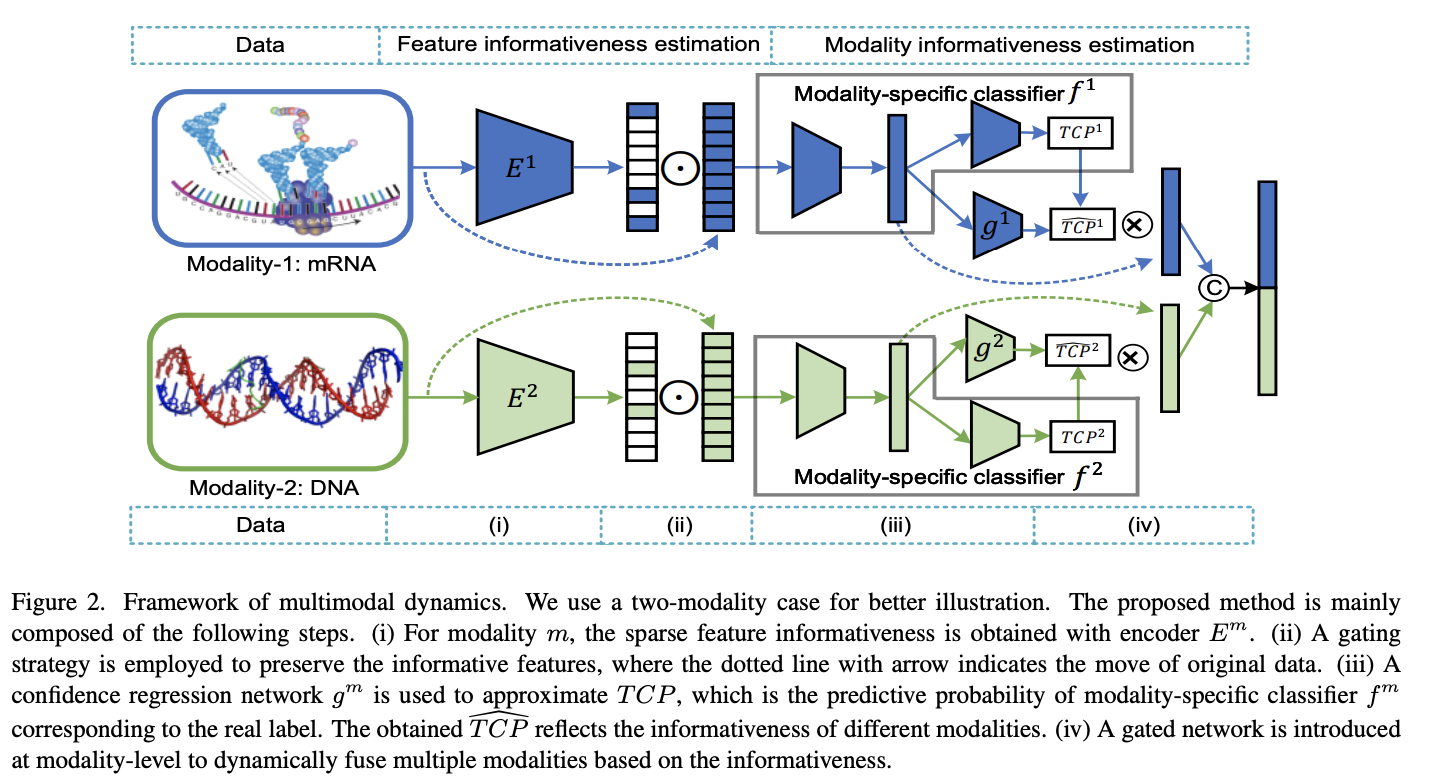
Notation
- \(N\): Number of samples
- \(M\): Number of modalities
- \(x_n^m \in \mathbb{R}^{d_m}\): n-th sample m-th modality (High-Dimensional)
- \(y_n \in \mathbb{R}^K\): n-th label
- \(\{\{x_n^m \}_{m=1}^M, y_n\}_{n=1}^N\): multimodal dataset (i.i.d)
위와같이 정의되어있을때, 기본적인 neural network는 \(f: \{x^m\}_{m=1}^M \rightarrow y\)이다.
Feature-level Dynamics
해당 논문은 위에서 언급한 (3)의 문제를 해결하기 위하여 Feature Selection방법을 수행한다. 수행하는 이유에 대해서는 다음과 같이 언급하였다.
(i) retaining important features and removing redundant and noisy ones, thereby promoting the multimodal fusion; (ii) enhancing the explanation ability of the multimodal fusion
(1) important feature만 남기고 상관없거나 noisy인 feature는 제외한다. multimodal fusion에서 explanation을 높이도록 수행한다.
Feature-informativeness encoder.
- \(E^m: x^m \rightarrow w^m\): m-th encoder network
- \(w^m \in \mathbb{R}^{d_m}\): weiht(feature informativeness vector)
$$w^m = \sigma(E^m(x^m)) = [w_1^m, \ldots, w_{d_m}^m], \sigma: \text{sigmoid function}$$
$$ L_{l_0}^s = \sum_{m=1}^M \sum_{d=1}^{d_m}s_d^m, \text{with } s_d^m= \begin{cases} 1, & \mbox{if }w_d^m \neq 0 \\ 0, & \mbox{otherwise} \end{cases}$$
저자들은 Feature informativeness estimation을 위하여 위와 같은 과정으로 Feature Selectiond을 수행하였다.
(1) Encoder로서 Reconstructiond을 수행하였다. 해당 과정에서 Input과 Output의 Dimension은 동일하며, Layer가 1개이다.
(2) Encoder를 학습함과 동시에 \(L_{l_0}^s\)인 L0 norm을 사용하여 Output에 대한 Mask형식을 구하였다. 하지만, 해당 저자도 마찬가지로 L0 norm은 optimize하기 힘듦으로 approx된 L1 norm을 사용하였다.
위의 과정으로 인하여 대표되는 Feature몇몇을 선택할 수 있으며, 값이 비슷하거나 매우 작은 값으로서 이루워진 noisy data를 제거할 수 있다는 것을 알 수 있다.
개인적인 생각으로는 이러한 과정을 거치기 위해서는 Data preprocessing과정에서 min-max normalization이 필수라고 생각된다.
Modality-level Dynamics
해당 논문에서 설명하고 있는 Multi-modality의 문제점에 대하여 다음과 같이 언급하고 있다.
For multimodal data, the informativeness of a modality is basically not fixed for different samples [28, 49]. Therefore, it is crucial for multimodal classification to be aware of the informativeness variation with respect to different samples, which is related to whether the model can adapt to the quality variation of modalities.
개인적인 생각으로서, Bio Domain에서 Gene Expression에서 이상이 없어도, DNA Methlaytion에서 문제가 생길 수 있다. 즉, 하나의 Disease (Label)을 예측함에 있어서 각각의 환자별로 modality의 중요도가 다를 수 있다. 이러한 modality의 중요도를 측정하는 것을 “Modality informativeness estimation”의 부분이다.
Maximum class probability
- \(f^m: x_n^m \rightarrow y_n\): m-th classifier
- \(p^m(y|x^m) = [p_1^m, \ldots, p_K^m]\): predictive distribution (Softmax output)
위와 같이 일반적인 softmax output의 notationd을 정의하게 되면, Loss Function(NLL)은 아래와 같다.
$$L^{cls} = - \sum_{m=1}^M \sum_{k=1}^K y_k \text{log}(p_k^m)$$

Dynamical Multimodal Fusion
위에서 설명한 “Feature-level Dynamics”으로 인하여 feature-level informativenss(\(\{w^m\}_{m=1}^M\))과 “Modality-level Dynamics”으로 인하여 modality-level informativeness(\(\hat{TCP}^m = g^m(x^m)\))를 구할 수 있었다.
해당 논문은 이러한 2가지 값을 이용하여 최종적인 model prediction은 다음과 같이 구하였다.
- \(\tilde{x} =sx^m \odot w^m, \odot: \text{elment-wise multiplication}\): Feature에 Weight를 주어서 important feature의 값만 살리는 과정.
- \(h^m = f_1^m(\tilde{x})\): Important Feature -> Feature Extractor -> Output.
- \(\hat{TCP}^m = g^m(x^m)\): Modality Confidence.
- \(h = [\hat{TCP}^1h_1, \ldots, \hat{TCP}^mh_m], [.,.]: \text{concatenation}\): multimodal representation considering modality confidence.
- \(f: h \rightarrow y\): Additional classifier is trained with cross-entropy Loss (\(L_f\)).
$$L = \sum_{i=1}^N (L^f + \lambda_1 L_{l_1}^s + \lambda_2 L^{conf}), \lambda_1, \lambda_2: \text{hyperparameters}$$
Experimental Setup
- Datasets:
- BRCA: 875 samples, 5 classes (PAM50 subtype)
- LGG: 510 samples, 2 samples
- ROSMAP: 351 samples, 2 classes (AD or NC)
- KIPAN: 658 samples, 3 classes (kidney cancer type)
Quantitative Analysis
Multi-class classification & Binary classification
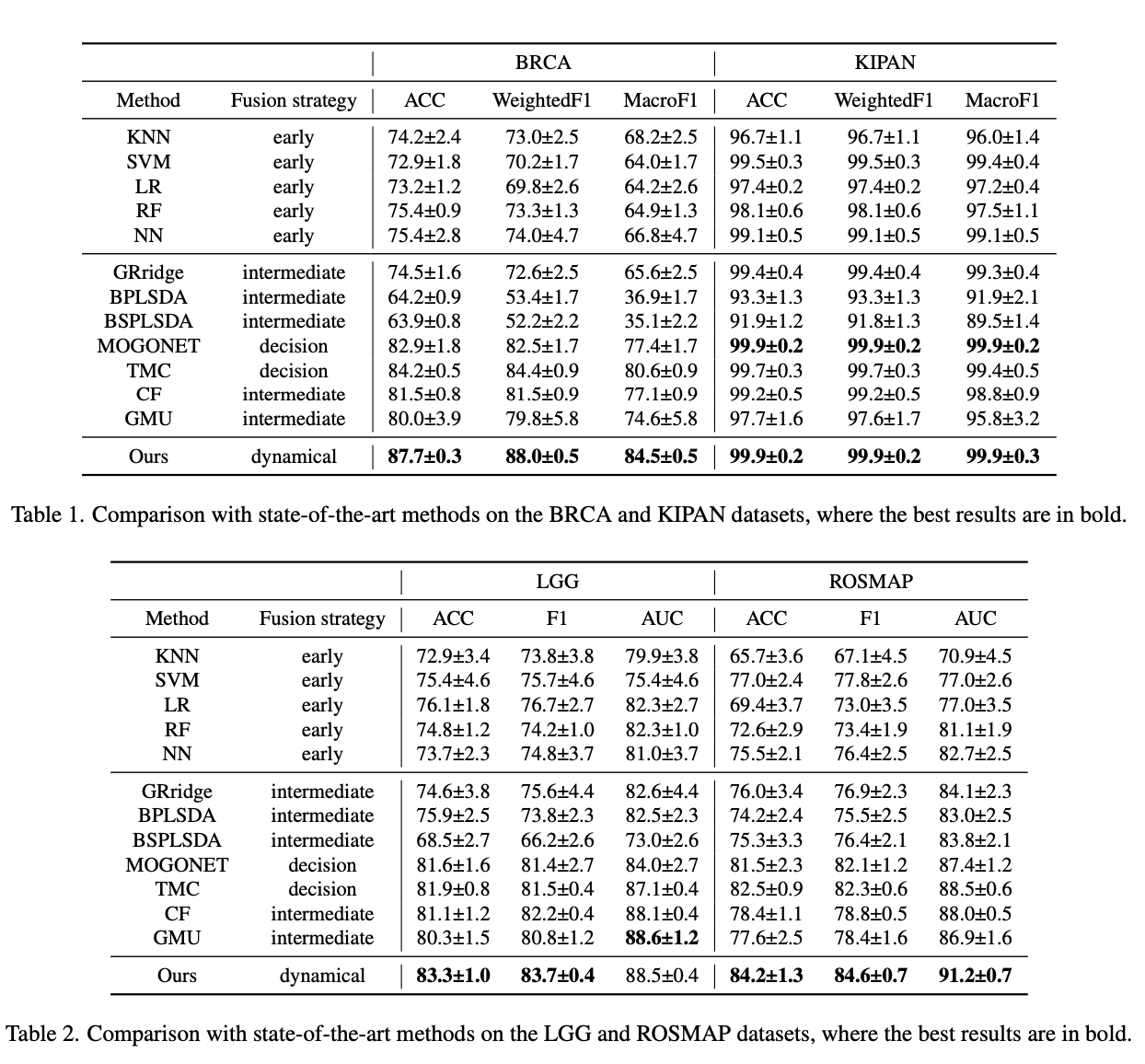
해당 논문에서 제시하는 방법은 Binary Classification뿐만 아니라 Multi-class classification에서도 모두 best performance를 보여주었다.
Ablation study
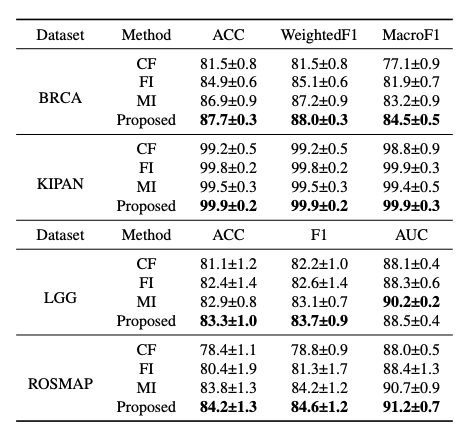
- concatenation of final multimodal representations (CF)
- sparse feature informativeness induced integration (FI)
- and modality informativeness induced integration (MI)
Ablation study결과를 살펴보게 되면, CF가 가장 많이 영향을 받고 각각의 FI와 MI또한 Performance에 영향을 주는 것을 알 수 있다. 특히, MI보다 FI가 효과적인 것을 알 수 있다. (CF를 제외하는 것은 어떻게 prediction하는지는 잘 모르겠습니다.)
Qualitative Analysis
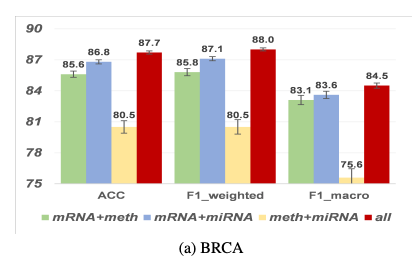
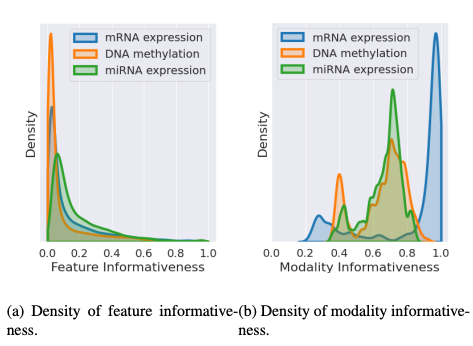
먼저 위쪽 Figure를 살펴보게 되면, BRCA Dataset에서 Modality를 하나씩 제거하면서 Performance의 변화를 살펴보았다. 해당 결과 Performance에 영향을 많이 미치는 Modality의 순위는 다음과 같다.
- mRNA
- miRNA
- meth
이러한 위에서 Performance에 영향을 많이 미치는 순으로 Modality informativeness density plot또한 같은 결과를 보여주었다.
Conclusion
해당 논문에서는 기존의 OOD sample을 제거하는데만 사용되었던 TCP를 활용하여, Modality의 Confidence를 적용하였다. 간단한 아이디어 추가와 이를 보여주기 위한 실험 설계를 잘 한 논문으로 생각된다.
PytorchCode
Model
- self.FeatureInforEncoder: Feature-informativeness encoder(\(E^m(\cdot)\))
- self.TCPConfidenceLayer: Multimodal confidence (\(g^m(\cdot)\))
- self.TCPClassifierLayer: m-th modality Classifier
- self.FeatureEncoder: Feature Extractor (\(f^m(\cdot)\))
- self.MMClasifier: Classifier (\(h \rightarrow y\))
Forward s
- torch.sigmoid(self.FeatureInforEncoder [view](data_list [view])):Feature-level informativeness (\(w^m = \sigma(E^m (x^m))\))
- feature [view] = data_list [view] * FeatureInfo [view]:Feature에 Weight를 주어서 important feature의 값만 살리는 과정 (\(\tilde{x} = x^m \odot w^m\))
- feature [view] = self.FeatureEncoder [view](feature[view]): Important Feature -> Feature Extractor -> Output(\(h^m = f_1^m(\tilde{x})\))
- TCPLogit [view] = self.TCPClassifierLayer [view](feature [view]): (\(TCP^m = y \cdot p^m(y|x^m)\))
- TCPConfidence [view] = self.TCPConfidenceLayer [view](feature [view]):Modality Confidence (\(\hat{TCP}^m = g^m(x^m)\))
- feature [view] = feature [view] * TCPConfidence [view]: (\(\hat{TCP}^mh_m\))
- MMfeature = torch.cat([i for i in feature.values()], dim=1): multimodal representation considering modality confidence (\(h = [\hat{TCP}^1h_1, \ldots, \hat{TCP}^mh_m]\))
- MMlogit = self.MMClasifier(MMfeature):Additional classifier is trained with cross-entropy Loss (\(f: h \rightarrow y\))
- MMLoss = torch.mean(criterion(MMlogit, label)):Cross-entropy Loss (\(L^f\))
- torch.mean(FeatureInfo [view]): \(L_{l_1}^s = \sum_{m=1}^M \|w^m\|_1\)
- confidence_loss = torch.mean(F.mse_loss(TCPConfidence [view].view(-1), p_target)+criterion(TCPLogit [view], label)): \(L^{conf} = \sum_{m=1}^M (\hat{TCP}^m - TCP^m)^2 + L^{cls}\)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
class LinearLayer(nn.Module):
def __init__(self, in_dim, out_dim):
super().__init__()
self.clf = nn.Sequential(nn.Linear(in_dim, out_dim))
self.clf.apply(xavier_init)
def forward(self, x):
x = self.clf(x)
return x
class MMDynamic(nn.Module):
def __init__(self, in_dim, hidden_dim, num_class, dropout):
super().__init__()
self.views = len(in_dim)
self.classes = num_class
self.dropout = dropout
self.FeatureInforEncoder = nn.ModuleList([LinearLayer(in_dim[view], in_dim[view]) for view in range(self.views)])
self.TCPConfidenceLayer = nn.ModuleList([LinearLayer(hidden_dim[0], 1) for _ in range(self.views)])
self.TCPClassifierLayer = nn.ModuleList([LinearLayer(hidden_dim[0], num_class) for _ in range(self.views)])
self.FeatureEncoder = nn.ModuleList([LinearLayer(in_dim[view], hidden_dim[0]) for view in range(self.views)])
self.MMClasifier = []
for layer in range(1, len(hidden_dim)-1):
self.MMClasifier.append(LinearLayer(self.views*hidden_dim[0], hidden_dim[layer]))
self.MMClasifier.append(nn.ReLU())
self.MMClasifier.append(nn.Dropout(p=dropout))
if len(self.MMClasifier):
self.MMClasifier.append(LinearLayer(hidden_dim[-1], num_class))
else:
self.MMClasifier.append(LinearLayer(self.views*hidden_dim[-1], num_class))
self.MMClasifier = nn.Sequential(*self.MMClasifier)
def forward(self, data_list, label=None, infer=False):
criterion = torch.nn.CrossEntropyLoss(reduction='none')
FeatureInfo, feature, TCPLogit, TCPConfidence = dict(), dict(), dict(), dict()
for view in range(self.views):
FeatureInfo[view] = torch.sigmoid(self.FeatureInforEncoder[view](data_list[view]))
feature[view] = data_list[view] * FeatureInfo[view]
feature[view] = self.FeatureEncoder[view](feature[view])
feature[view] = F.relu(feature[view])
feature[view] = F.dropout(feature[view], self.dropout, training=self.training)
TCPLogit[view] = self.TCPClassifierLayer[view](feature[view])
TCPConfidence[view] = self.TCPConfidenceLayer[view](feature[view])
feature[view] = feature[view] * TCPConfidence[view]
MMfeature = torch.cat([i for i in feature.values()], dim=1)
MMlogit = self.MMClasifier(MMfeature)
if infer:
return MMlogit
MMLoss = torch.mean(criterion(MMlogit, label))
for view in range(self.views):
MMLoss = MMLoss+torch.mean(FeatureInfo[view])
pred = F.softmax(TCPLogit[view], dim=1)
p_target = torch.gather(input=pred, dim=1, index=label.unsqueeze(dim=1)).view(-1)
confidence_loss = torch.mean(F.mse_loss(TCPConfidence[view].view(-1), p_target)+criterion(TCPLogit[view], label))
MMLoss = MMLoss+confidence_loss
return MMLoss, MMlogit
def infer(self, data_list):
MMlogit = self.forward(data_list, infer=True)
return MMlogit
Hyperparameter
1
2
3
4
5
6
7
8
9
10
11
12
13
if 'BRCA' in data_folder:
hidden_dim = [500]
num_epoch = 2500
lr = 1e-4
step_size = 500
num_class = 5
elif 'ROSMAP' in data_folder:
hidden_dim = [300]
num_epoch = 1000
lr = 1e-4
step_size = 500
num_class = 2
해당 Code에서 이해되지 않은 점은 다음과 같이 2가지 였다.
- Validation set을 따로 지정하지 않고 Train Test로만 나누었다. -> Validation이 따로 존재하지 않아서 Hyperparameter를 찾는 과정이 존재하지 않는다.
- Validation set이 따로 존재하지 않으므로 Early-Stopping도 진행하지 못하였다.

Leave a comment